O Hospital Israelita Albert Einstein, de São Paulo, desenvolveu o primeiro teste do mundo de diagnóstico do novo coronavírus baseado em Sequenciamento de Nova Geração (Next Generation Sequencing – NGS) com 100% de especificidade – ou seja, não apresenta casos de falso-positivo.
Sua precisão é equivalente à apresentada pelo método convencional, o RT-PCR, porém com a vantagem de permitir a realização simultânea de até 1536 amostras, totalizando um volume de processamento 16 vezes maior do que o possível pelo método RT-PCR, tido como padrão ouro. Protegida intelectualmente por meio do Sistema Internacional de Patentes nos Estados Unidos, a tecnologia representa um grande avanço no combate à pandemia da covid-19, doença causada pelo novo coronavírus, o Sars-Cov-2.
O novo recurso surge como uma alternativa viável de adoção de testagem diagnóstica em massa, ação vital para a tomada de medidas imediatas de tratamento, previsão da demanda para o sistema de saúde e controle da expansão dos casos. O que há disponível para testagem em massa são os exames sorológicos, conhecidos como testes rápidos.
No entanto, eles detectam anticorpos produzidos pelo organismo em resposta à infecção e só podem ser observados em média 14 dias após a contaminação. Por isto, são apenas para triagem e possuem taxas aproximadas de 30% de falsos-negativos.
O teste criado pelo Einstein, ao contrário, identifica a presença do vírus, funcionando como instrumento diagnóstico a ser usado desde o primeiro dia de infecção, da mesma forma que o RT-PCR. “A nova tecnologia amplia a capacidade mundial de diagnóstico, início rápido de tratamento e de isolamento dos doentes e contactantes, contribuindo, desta maneira, para o controle da expansão da pandemia”, afirma o médico Sidney Klajner, presidente da Sociedade Beneficente Israelita Brasileira Albert Einstein.
O teste é baseado na tecnologia de Sequenciamento de Nova Geração (Next Generation Sequencing – NGS), que consiste na leitura de pequenos fragmentos de DNA para a identificação de doenças ou mutações genéticas. A grande inovação desenvolvida pelos pesquisadores do Einstein foi ter adaptado o método para detectar RNA, a outra molécula biológica que, junto com o DNA, compõe o material genético de todos os seres vivos.
Como diversos tipos de vírus, o Sars-Cov-2 possui apenas RNA. “Até agora, a única forma de registrar sua presença dentro das células humanas era pela técnica RT-PCR”, explica o patologista João Renato Rebello Pinho, coordenador do Laboratório de Técnicas Especiais do Hospital Israelita Albert Einstein.
O exame foi desenvolvido seguindo boas práticas e recomendações de instituições respeitadas como o Centro de Controle e Prevenção de Doenças (CDC), dos Estados Unidos, e a Food and Drug Administration (FDA), a agência americana responsável pela aprovação de medicamentos e tecnologias em saúde.
As primeiras validações demonstram alta capacidade (90%) de identificar corretamente os indivíduos que contraíram a doença. Elas foram executadas usando amostras testadas na rotina habitual do laboratório do Einstein.
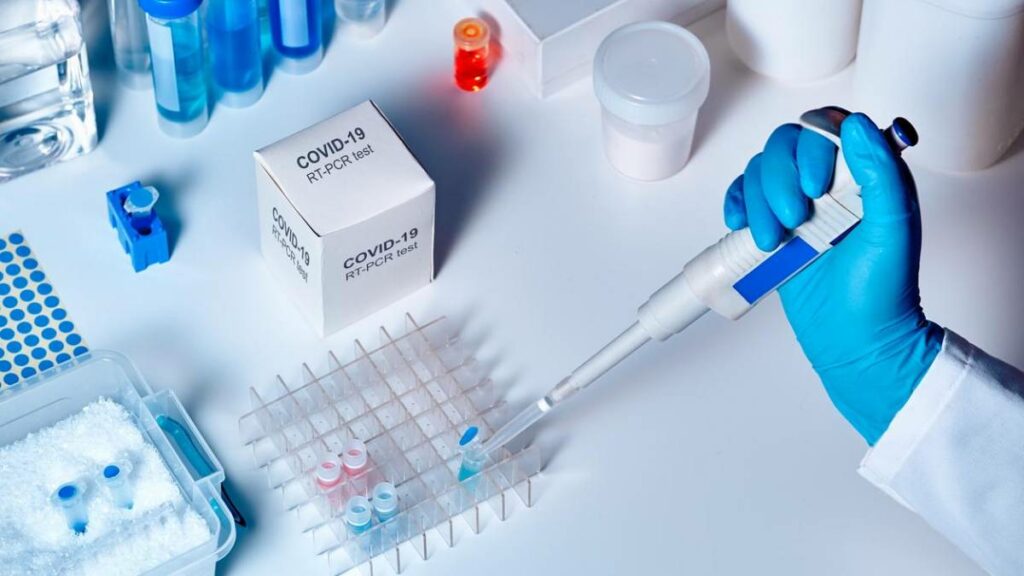
A coleta de amostra para detecção do vírus é realizada por meio de cotonetes estéreis (chamados de swab) em contato com a região nasal ou saliva. Posteriormente, a amostra é preparada de acordo com protocolos específicos desenvolvidos pelo hospital.
Por fim, a análise dos resultados é realizada por meio da plataforma de bioinformática Varstation, também criada pelo Departamento de Inovação do Einstein e comercializada para outras empresas. “O resultado fica pronto em até três dias, mas já estamos trabalhando para reduzir significativamente este prazo”, explica o bioinformata Murilo Cervato, gerente de Inovação e Ciência de Dados da organização e CEO da Varstation. Todo o processo está patenteado.
O teste deverá estar disponível para a rotina de operação diagnóstica no Einstein até o início de junho. Trata-se, portanto, de outra vantagem do exame criado no hospital, já que amplia de maneira significativa a capacidade de testagem no Einstein e, potencialmente, a de outros laboratórios semelhantes no Brasil e no mundo.
A adoção da metodologia patenteada pelo Einstein tem grande potencial de ser adotada em larga escala, uma vez que há grande disponibilidade de produção em laboratórios de NGS, que atualmente se encontram, na maior parte dos casos, com baixo nível de utilização em função da pandemia.
O desenvolvimento do teste levou cerca de dois meses. “O feito da equipe do Einstein, executado em tão pouco tempo, é resultado do grande investimento da organização em suas áreas de pesquisa, inovação e empreendedorismo”, afirma o engenheiro Claudio Terra, diretor de Inovação e Transformação Digital da Sociedade Beneficente Israelita Brasileira Albert Einstein.
O passo a passo do novo teste genético que diagnostica o Sars-Cov-2
O exame é baseado na tecnologia de Sequenciamento de Nova Geração (Next Generation Sequencing – NGS), que consiste na leitura de pequenos fragmentos de DNA para a identificação de doenças ou mutações genéticas
Coleta da amostra: Ela é feita por meio de hastes flexíveis estéreis chamadas de swab em contato com a região nasal ou saliva, como no RT-PCR.
Extração do material genético: Em seguida, parte-se para a obtenção do material genético do vírus, o RNA, um processo que leva menos de 30 minutos. Para isso, são usados reagentes, de acordo com o protocolo desenvolvido pelo Einstein. O protocolo não se vale dos kits convencionais de extração de RNA utilizados para o RT-PCR. Dessa forma, não concorre com o acesso aos mesmos insumos necessários para o RT-PCR.
Amplificação do material genético: Os especialistas recorrem a uma enzima, a transcriptase reversa, para transformar o RNA do vírus em DNA complementar, também chamado de cDNA. É uma fita de DNA obtida no momento da transcrição das informações genéticas, como se fosse uma cópia do original.
Preparo de biblioteca de sequenciamento: Nesta etapa, entram os primers universais – em outras palavras, o primer é uma fita simples de DNA que auxilia na amplificação do material genético de interesse em 100 milhões de vezes.
Sequenciamento do DNA: Aqui, determina-se a sequência de letras que, encadeadas uma depois da outra, compõe o genoma completo do vírus.
Análise dos dados: É feita em uma ferramenta de bioinformática, o Varstation, que também foi desenvolvida pelo departamento de Inovação do Einstein. Utiliza-se um pipeline, isto é, uma sequência de instruções computacionais. Este pipeline é composto por um algoritmo de inteligência artificial capaz de identificar os pacientes e os respectivos resultados – se é positivo ou negativo –, que saem em até 72 horas após a coleta do material biológico. É possível analisar 1536 amostras ao mesmo tempo, contra as 96 do RT-PCR.